您现在的位置: 6165cc金沙总站检测中心» 学院动态» 【科研新进展】学院姜雨教授团队反刍动物多组学数据库2.0版本开源发布
【科研新进展】学院姜雨教授团队反刍动物多组学数据库2.0版本开源发布
发布日期:2021-10-14 浏览次数:
10月14日,学院反刍动物遗传与进化研究中心姜雨教授团队在国际著名杂志《Nucleic Acids Research》发表题为“RGD v2.0: a major update of the ruminant functional and evolutionary genomics database”的研究成果。该研究构建了国际上最大规模的反刍动物功能与进化基因组学数据库(http://animal.nwsuaf.edu.cn/RGD),是继2019年反刍项目Science系列文章发布后的再次更新。博士后付玮玮和硕士生王睿为论文的共同第一作者,姜雨教授为论文的通讯作者。
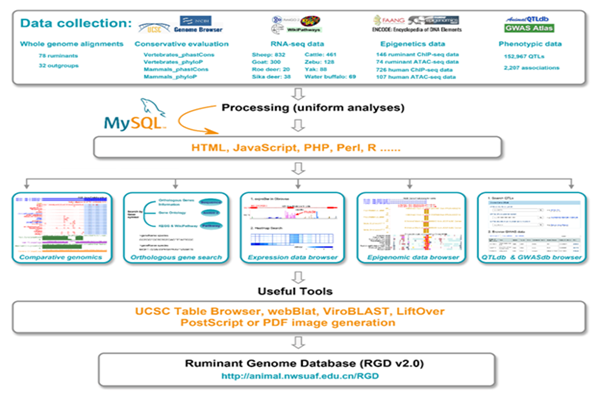
反刍动物功能与进化基因组学数据库(RGD)的内容和构建流程
在动物育种中,如何确定靶标基因和位点是制约反刍家畜缓慢育种的主要原因,单一组学的分析往往无法准确解释基因型与表型间的因果关系。而现有资源库大多只关注某一物种的多组学数据,难以实现不同库间的有效整合和标准化利用,从而形成信息屏障,无法有效进行基因型到表型的准确推测。本研究通过多算法多层次的组学数据全面解析和注释了反刍动物功能元件,为家畜育种位点筛选提供了重要资源。目前数据库中包含了基于牛、绵羊和山羊基因组为参考构建的三套坐标系的110个物种(78个反刍物种和32个具有代表性节点的外群物种)的基因组共线性比对;直系同源基因的鉴定和注释;从宏进化角度鉴定到的保守元件;利用1053 个表观数据拿到的近一百万个顺式调控元件;1936个转录组数据分析的基因表达数据;以及收集到的155174条表型数据。此外,数据库还提供了反刍物种常用家畜的基因组坐标转换工具liftOver,方便不同基因组版本之间进行位点转换,甚至包括人的基因组功能元件数据的迁移。
反刍亚目是最成功的哺乳动物类群之一,总体重占据所有哺乳动物的50%以上,其占据了跨度大的生态位(如适应极寒、极热环境),而且包括了多个畜种,如黄牛、牦牛、水牛、绵羊、山羊和驯鹿等。随着高通量测序技术的发展,反刍动物的多组学数据得到快速积累,三代组装技术使得反刍物种的基因组有了质的提升。本团队通过收集公共数据库中几乎所有与反刍动物相关的基因组、转录组、表观组和表型组的数据,利用比较基因组共线性比对实现反刍物种内的多组学数据共注释,形成详细的功能元件信息,并通过内嵌的基因组“浏览器”、丰富的数据检索模块和展示工具实现不同组学数据的直观比对。该研究论文还列举了一系列基因来阐述数据库的功能。如,与牛体高相关的 PLAG1 基因,与绵羊繁殖相关的 FSHR 基因,以及与山羊绒毛相关的 FGF5 基因,这些基因的关键变异位点均落在反刍动物启动子和增强子相关的功能元件上,表明数据库可以通过功能元件信息快速锁定经济性状相关的关键变异。
此外,反刍动物遗传与进化研究团队还开发了牛的变异数据库(http://animal.nwsuaf.edu.cn/BosVar)和山羊的变异数据库(http://animal.nwsuaf.edu.cn/GoatVar),收纳了世界家牛和家羊不同品种的大量重测序数据,用户可以以曼哈顿图或折线图查看受选择区域,以地图映射的方式查看并下载变异位点在不同群体中的频率变化。用户通过将牛羊变异数据库与本次研究开发的反刍动物功能元件配合使用,可以有效锁定特定群体(或品种)的选择信号区域,筛选具有明显频率差,并具有功能元件注释的关键变异位点,从而大幅度缩小候选位点的范围,减少下游实验验证的工作量。
该研究得到国家自然科学基金优秀青年基金(31822052)的资助,以及西北农林科技大学高性能计算平台和双一流学科群的支持。
原文链接:https://academic.oup.com/nar/advance-article-abstract/doi/10.1093/nar/gkab887/6395343
