您现在的位置: 6165cc金沙总站检测中心» 学院动态» 【科研新进展】我院姜雨教授团队发布迄今最为完整的山羊参考基因组
【科研新进展】我院姜雨教授团队发布迄今最为完整的山羊参考基因组
作者:樵淑银 发布日期:2021-09-14 浏览次数:
近日,姜雨教授团队在动物遗传育种领域Top期刊Genetics Selection Evolution发表了题为“A near complete genome for goat genetic and genomic research”的研究论文,李冉副教授和研究生杨鹏、代学雷为本论文共同第一作者,姜雨教授为本论文的通讯作者,西北农林科技大学为第一单位。
利用高深度Pacbio测序和Hi-C辅助组装技术报道了首个萨能奶山羊的高质量参考基因组Saanen_v1,该基因组序列已在NCBI数据库公布(GCA_015443085.1),其组装连续性指标contigN50为46Mb,是目前组装连续性最好的动物基因组之一,全基因组断点数只有169个,远低于牛参考基因组的386个、猪参考基因组的506个、以及人的875个断点。对29条山羊常染色体中的20条同时组装出了端粒序列和着丝粒序列,向完整组装山羊基因组又前进了一大步。
姜雨教授团队长期开展羊基因组解析完善和泛基因组构建工作,作为主要完成人之一于2013年报道了第一个山羊的参考基因组CHIR_1.0。2016年,美国农业部科研人员利用Pacbio测序技术于组装了山羊的新参考基因组ARS1,整体完整度有显著提高,但由于测序深度在X染色体只有35x,组装的X染色体仍很不完整,且未能组装出Y染色体,给后续研究带来了不便。萨能奶山羊原产于瑞士,是世界上最优秀的奶山羊品种之一,对关中奶山羊和西农萨能奶山羊的选育均有重要的贡献。本研究选取一只萨能奶山羊种公羊,利用最新的三代Pacbio测序技术和Hi-C辅助组装进行了高深度测序,三代测序深度达到了117x,并最终组装出了萨能奶山羊的参考基因组Saanen_v1,特别是包含了目前最为完整的X染色体序列和首个Y染色体序列。不同评估指标表明该基因组的完整度和连续性远优于参考基因组ARS1的组装水平,并且纠正了当前参考基因组ARS1的8个大于1Mb的基因组组装错误区域,并发现山羊GoatSNP50芯片中4.4%的SNP探针基因组定位信息不正确,为新的山羊育种芯片设计提供了重要参考。
萨能奶山羊高质量基因组的解析将进一步促进新一代山羊育种芯片的研发进程,对山羊的遗传育种研究将有重要的推动作用。
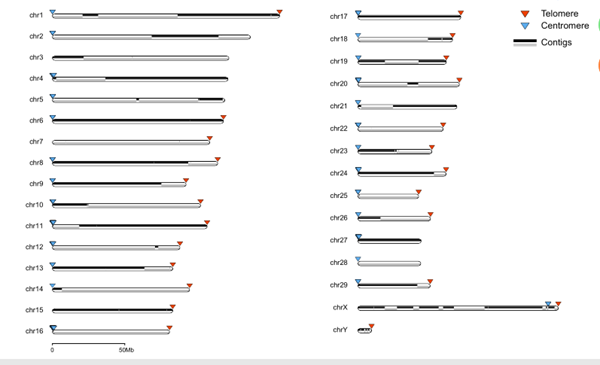
图1 萨能奶山羊基因组染色体
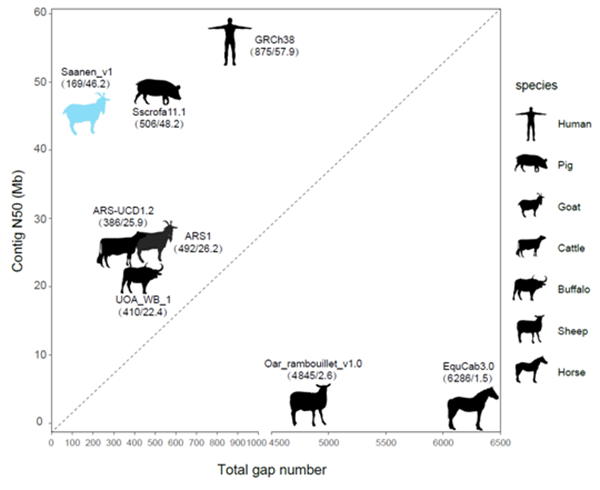
图2 各种哺乳动物基因组组装质量比较(断点数量,平均组装长度)
论文链接:https://gsejournal.biomedcentral.com/articles/10.1186/s12711-021-00668-5
